mRNA检测服务
质量控制作为药物开发最重要的一环,对于药物成分、杂质等的分析是药物开发过程中的重中之重。瀚海新酶提供mRNA药物质检服务




帽结构及加帽率检测原理
mRNA的结构主要包括 5'端帽子( cap,简称帽子) 、5'端非翻译区(untranslated region,UTR)、开放阅读框( open reading frame,ORF) 、3'端非翻译区和多聚腺苷酸尾巴 [Poly( A) ]等结构。成熟的真核生物 mRNA的 5'端都有1个甲基鸟苷帽( m7G) 结构。加帽能够帮助mRNA 被顺利转运出细胞核,增加 mRNA 稳定性,防止其被核糖核酸酶降解,对蛋白的表达至关重要。

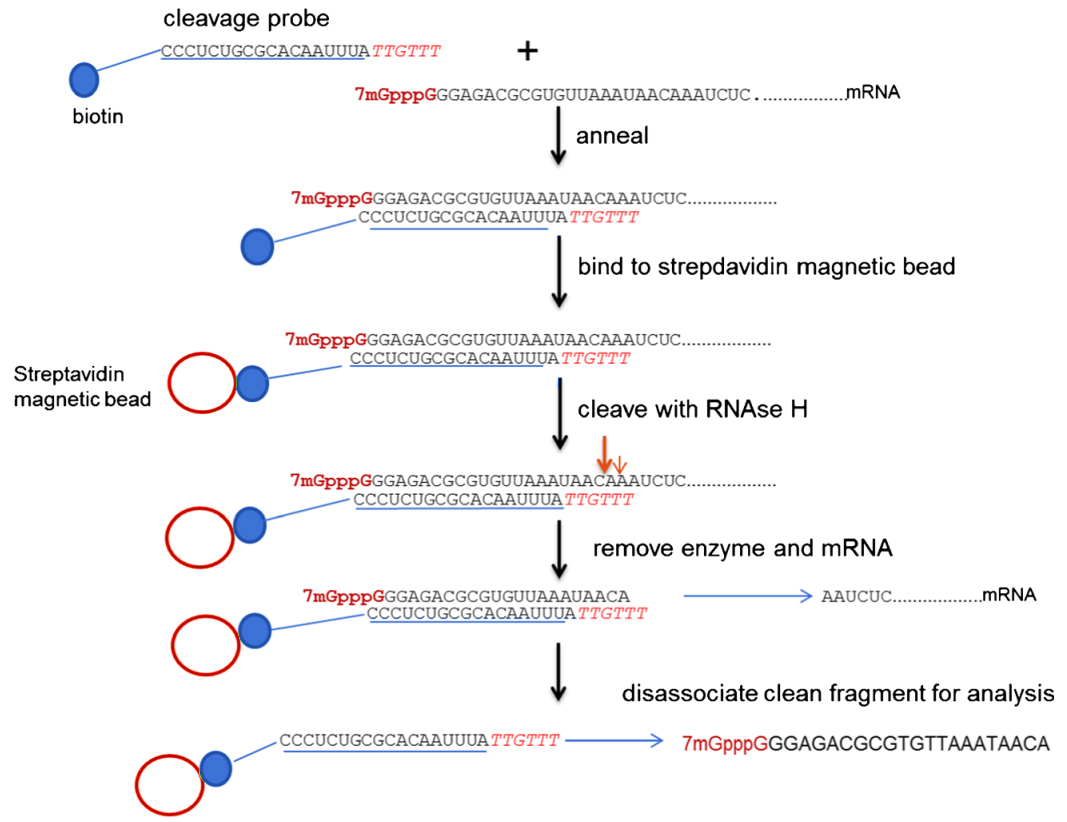
加帽率检测目前采用的主要手段是先合成与5'UTR区前50nt左右相配对的含有生物素的探针,让探针和mRNA结合,然后用RNase H进行酶切,利用链霉亲和素磁珠特异性的富集带有帽子结构序列的探针,最后将探针上的含有帽子结构的序列洗脱下来,进入到LC-MS进行分析。
Poly(A)结构及Poly(A)分布检测原理
真核生物的mRNA通常在其3'端有一段腺苷,根据物种的不同,其长度从20到200多个碱基不等。最近有一些研究表明mRNA较长的尾巴可以提高翻译效率,提供mRNA的稳定性,并影响免疫反应。
目前主要采用RNase T1酶进行酶切,酶切后的Poly(A)片段用Oligo DT填料进行富集,将富集在填料上的Poly(A)片段洗脱下来后进入到 LC-MS进行分析。
服务类别 | 用时(工作日) | 交付 | 标准 |
加帽率检测探针设计合成 | 5 | 探针 | 根据客户需求确定 |
加帽率检测样品前处理 | 2 | 前处理样品 | |
加帽率检测样品上机检测 | 3-10 | 检测报告 | |
Poly(A)分布检测样品前处理 | 2 | 前处理样品 | |
Poly(A)分布检测样品上机检测 | 3-10 | 检测报告 |

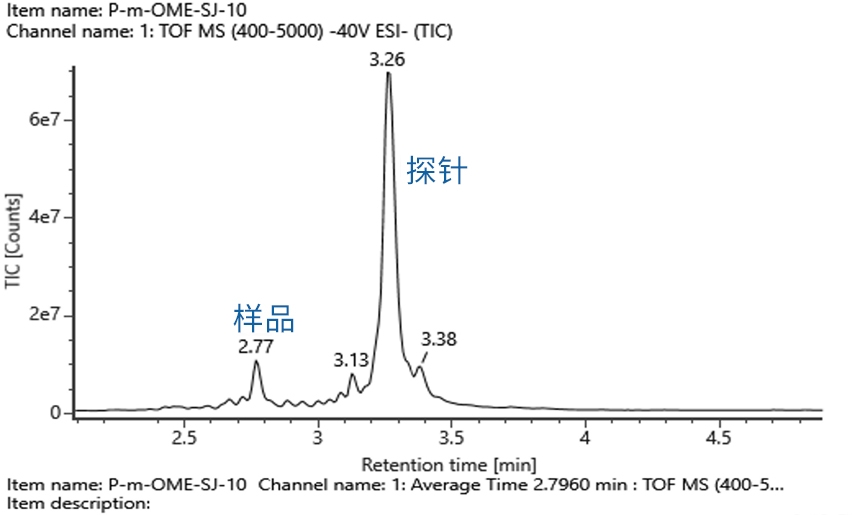
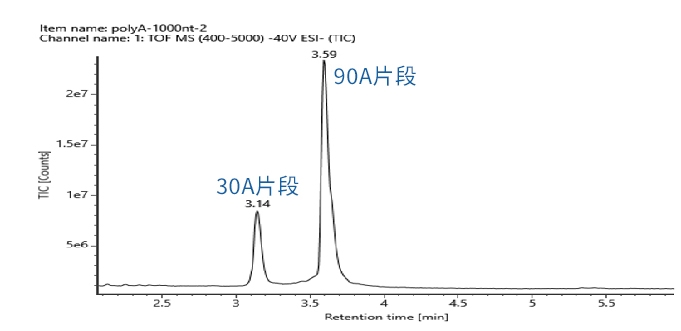
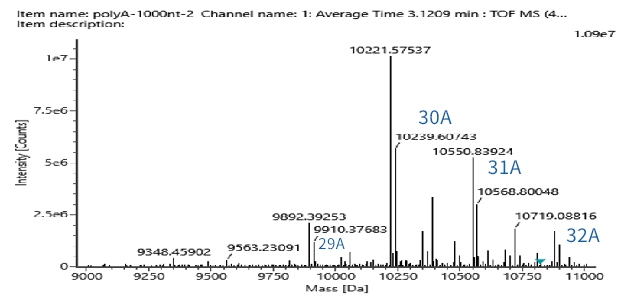

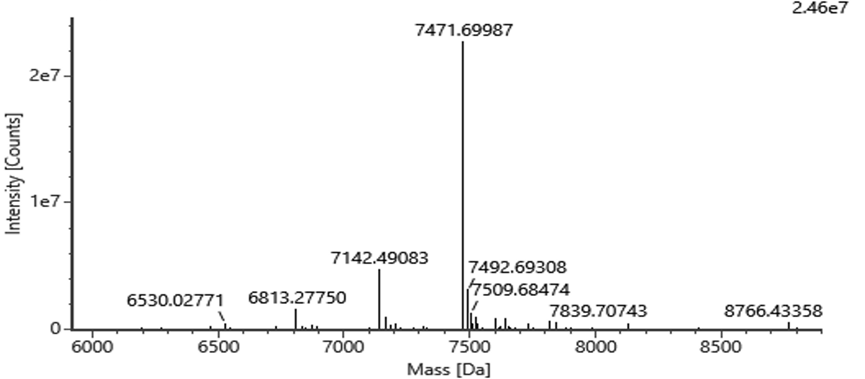
Name | Response | Observed mass (Da) | Expected mass (Da) | Mass error (mDa) | Mass error (ppm) | Cap1(%) |
Monphos | 38908 | 7004.38 | 7004.29 | 88.1 | 12.6 | 95.1% |
Triphos | 1052878 | 7164.47 | 7164.25 | 223.1 | 31.1 | |
Triphos_a | 506256 | 6835.27 | 6835.05 | 225.3 | 33.0 | |
Diphos_a | 101498 | 6755.32 | 6755.07 | 256.5 | 38.0 | |
cap1 | 23880426 | 7471.69 | 7471.55 | 143.2 | 19.2 | |
cap1_gaa | 1924043 | 6468.05 | 6467.93 | 125.6 | 19.4 | |
cap1_a | 5077712 | 7142.48 | 7142.34 | 138.5 | 19.4 | |
cap1_aa | 2301145 | 6813.27 | 6813.13 | 139.1 | 20.4 |

